铜绿假单胞菌的基因组耐药性与表型易感性之间的关系
铜绿假单胞菌(Pseudomonas aeruginosa, P.ae)是囊性纤维化(CF)患者发病和死亡的主要原因,也是从重症监护病房获得的感染中分离出来的第三大常见细菌。因其存在的复杂的耐药机制,P.ae引起的感染难以被有效治疗,成为临床的“难对付”的菌。此外,流行病学研究显示该菌对愈来愈多的抗生素耐药,包括“第四代”头孢菌素,而仅有多粘菌素和阿米卡星对这种多重耐药P.ae保有良好的杀伤活性。因而,为应对不断上升的细菌耐药性发生率,临床上需要一种快速且可靠的方法以实现抗生素药物敏感性的预测,以确保进行正确的治疗。基于PCR、微阵列的检测方法在定义革兰氏阴性菌耐药性方面得到了较为广泛的应用,但这样的方法不能应对细菌不断更新的耐药机制——潜在候选基因和检测的覆盖范围不足。相比之下,NGS是一个可靠的方案,在肠球菌、沙门氏菌、大肠杆菌和肺炎克雷伯菌的耐药表型预测上已得到了证明。但鲜有研究将NGS作为一种识别耐药性相关因素的方法,并建立其预测结果与传统方法获得的药敏结果的相关性。基于此背景,Veronica N. Kos等人对2003年至2012年间收集的来自不同地理位置和临床感染的390株P.ae进行测序,开展全基因组分析,以确定该病原耐药机制的多样性和分布;再将分析结果用于耐药性预测,并对预测结果作出评价。
作者从NCBI下载了15株P.ae的完整基因组作为参考(PAO1),将测序得到的390株P.ae基因组序列与之比对,开放阅读框(Open Reading Frame,ORF)相似度高于90%的用于系统发育树构建。系统发育分析和基因组大小的评估表明,研究的菌株是P.ae的异质性群体(图1)。研究对菌株基因组进行了多位点序列分型(Multilocus sequence typing, MLST),结果显示了某些克隆株仅出现在特定地理位置,这反映了环境P.ae的传播与患者之间存在一定关联。由于AR基因的水平获得通常与整合子相关,因而作者展开了一项旨在确定假定的整合子结构的研究,特别是它们相关的β-内酰胺酶。研究在92株分离株中检测到水平获得性β-内酰胺酶基因,其中54株携带1个(除了染色体ampC和blaOXA-50),32株携带2个,6株含有3个(图2A)。另外,还观察到序列分型ST235和ST111携带了大量β-内酰胺酶基因(图2B),这在一定程度上显示了P.ae在医院环境中极强的获得耐药性的能力。随后,研究人员利用获得的基因组信息验证了基因型对临床分离株进行耐药/敏感分类的可行性。结果显示了P.ae耐药机制的多样性(图3A),NGS能够可靠地预测P.ae对美罗培南和左氧氟沙星的耐药性,但阿米卡星的耐药性预测效果并不理想(图3B、C),这可能是由于分离株所处的环境诱导了一种独特的表达相关机制。对此,研究人员表示,正在试图开发机器学习模型以识别这些分离株中产生阿米卡星耐药性的新序列。
总地来说,该研究证明了NGS在确定相关抗性基因方面的实用性,并强调了P.ae内抗性决定因素的多样性。这一信息对进一步设计治疗P.ae感染的诊断和治疗方法很有价值。
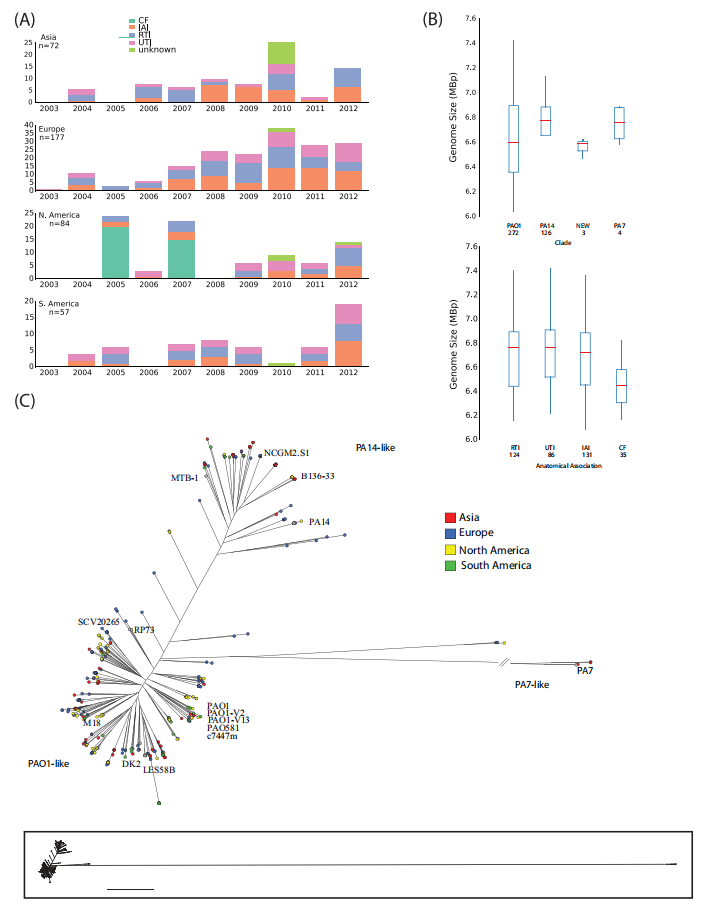
 图1 (A)获得P.ae分离株的时间、地理和部位来源。(B)箱形图描述了不同身体部位来源的P.ae的基因组大小。CF分离株作为一个单独的组。(C)405株P.ae的无根最大似然进化树。由与PAO1基因组比对得到的1278个基因的核心基因组的SNPs构建。
图1 (A)获得P.ae分离株的时间、地理和部位来源。(B)箱形图描述了不同身体部位来源的P.ae的基因组大小。CF分离株作为一个单独的组。(C)405株P.ae的无根最大似然进化树。由与PAO1基因组比对得到的1278个基因的核心基因组的SNPs构建。
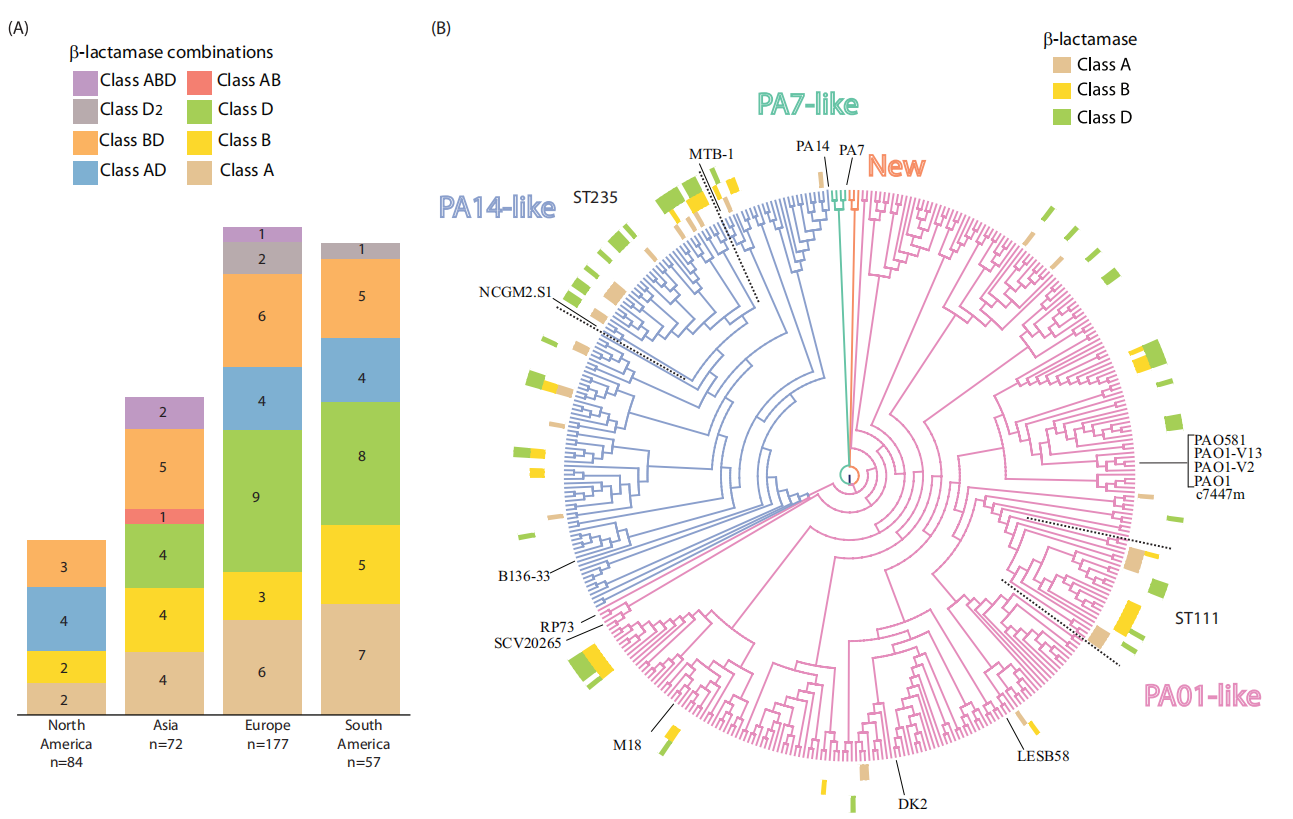
 图2 β-内酰胺酶含量汇总图。(A)390株P.ae中β-内酰胺酶在地理位置上的关联。(B)A类、B类和D类β-内酰胺酶的系统发育关联。图中显示了4种分支:PAO1.PA14.PA7以及尚未被定义的(新的)。在这些菌株中,序列分型ST235和ST111是富含β-内酰胺酶的两个最大类群。
图2 β-内酰胺酶含量汇总图。(A)390株P.ae中β-内酰胺酶在地理位置上的关联。(B)A类、B类和D类β-内酰胺酶的系统发育关联。图中显示了4种分支:PAO1.PA14.PA7以及尚未被定义的(新的)。在这些菌株中,序列分型ST235和ST111是富含β-内酰胺酶的两个最大类群。
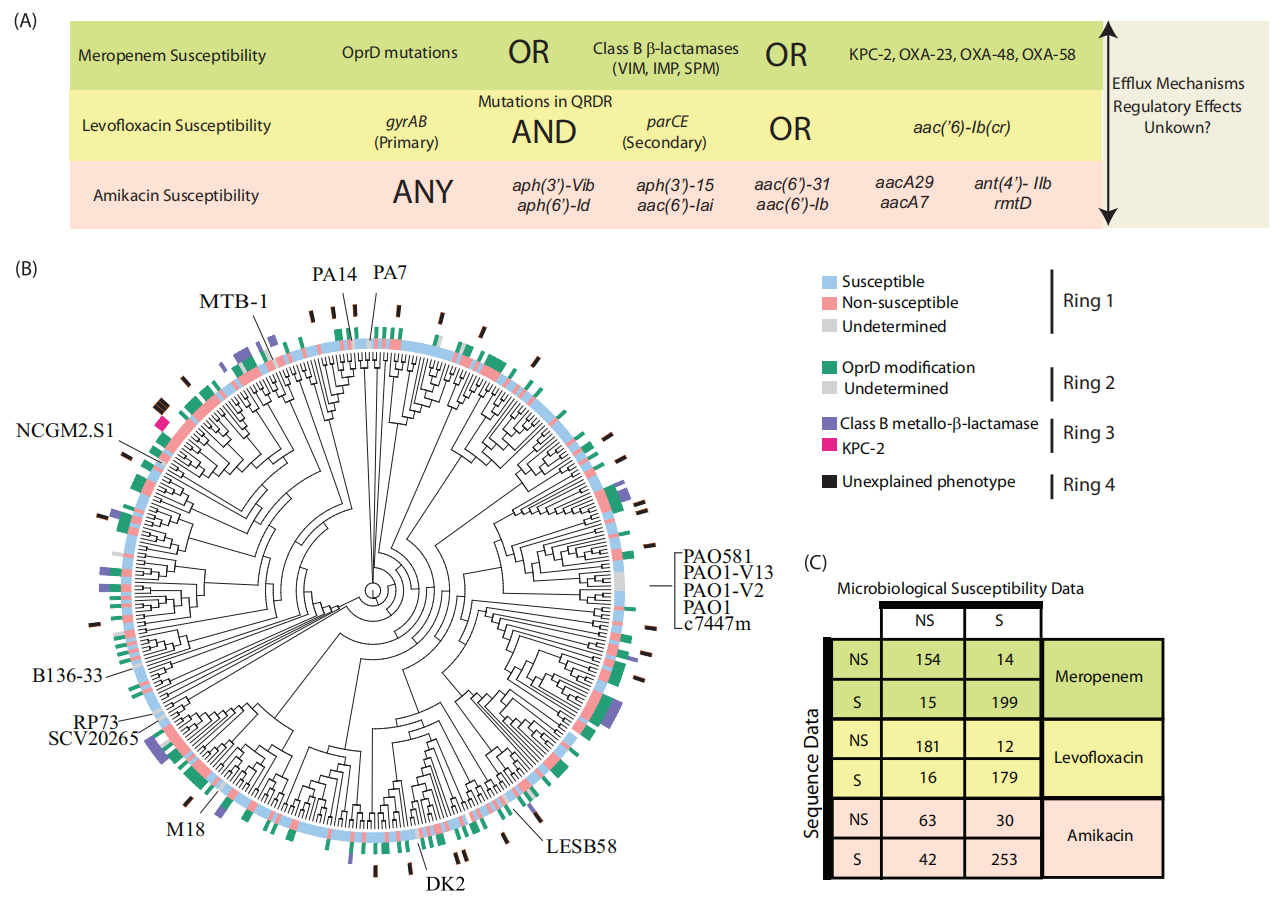
 图3 (A)基于遗传决定因素解释P.ae的遗传易感性的模型。美罗培南耐药性主要与孔蛋白OprD的序列修饰,β类内酰胺酶、碳青霉烯酶如KPC-2有关。左氧氟沙星耐药性主要通过gyrA/B和parC/E的QRDR内的突变组合来确定。阿米卡星耐药性则通过鉴定氨基糖苷耐药基因组合来确定。(B)系统发育树上菌株药敏结果与基于遗传因子辨别的药物敏感结果的比较。最内层的环(环1)显示了菌株表型药敏结果,浅红色代表耐药株,蓝色代表敏感株。参考菌株用灰色显示,仅用于定位。环2显示了具有OprD修饰的分离株(孔蛋白,绿色),及因序列覆盖不完全而无法推断OprD信息的分离株(灰色)。环3突出了携带B类MBL(紫色)和携带了碳青霉烯酶KPC-2(粉红色)的分离株。最外层的环(环4)则显示了在表型和基因型耐药性分析上不一致的菌株(黑色)。(C)基于遗传因素的P.ae耐药性预测与传统药敏测试结果的一致性评价(美罗培南、左氧氟沙星和阿米卡星)。
图3 (A)基于遗传决定因素解释P.ae的遗传易感性的模型。美罗培南耐药性主要与孔蛋白OprD的序列修饰,β类内酰胺酶、碳青霉烯酶如KPC-2有关。左氧氟沙星耐药性主要通过gyrA/B和parC/E的QRDR内的突变组合来确定。阿米卡星耐药性则通过鉴定氨基糖苷耐药基因组合来确定。(B)系统发育树上菌株药敏结果与基于遗传因子辨别的药物敏感结果的比较。最内层的环(环1)显示了菌株表型药敏结果,浅红色代表耐药株,蓝色代表敏感株。参考菌株用灰色显示,仅用于定位。环2显示了具有OprD修饰的分离株(孔蛋白,绿色),及因序列覆盖不完全而无法推断OprD信息的分离株(灰色)。环3突出了携带B类MBL(紫色)和携带了碳青霉烯酶KPC-2(粉红色)的分离株。最外层的环(环4)则显示了在表型和基因型耐药性分析上不一致的菌株(黑色)。(C)基于遗传因素的P.ae耐药性预测与传统药敏测试结果的一致性评价(美罗培南、左氧氟沙星和阿米卡星)。
论文DOI:10.1128/AAC.03954-14.
1、凡本网所有原始/编译文章及图片、图表的版权均属微生物安全与健康网所有,未经授权,禁止转载,如需转载,请联系取得授权后转载。
2、凡本网未注明"信息来源:(微生物安全与健康网)"的信息,均来源于网络,转载的目的在于传递更多的信息,仅供网友学习参考使用并不代表本网同意观点和对真实性负责,著作权及版权归原作者所有,转载无意侵犯版权,如有侵权,请速来函告知,我们将尽快处理。
3、转载请注明:文章转载自www.mbiosh.com
联系方式:020-87680942

