基于基因组数据的机器学习分析预测金黄色葡萄球菌抗菌素耐药表型的实用方法
随着测序价格的降低和测序速度的加快,将细菌的基因型和表型直接联系起来显得尤为重要。目前,许多研究都集中在抗菌素耐药性预测方面,一些研究仅预测了菌株是否耐药或敏感,而没有预测特异性MIC值,而其他人则基于已知耐药基因或单核苷酸多态性的存在与否以预测耐药性和易感性。然而,现有基础研究的更新相当缓慢。k-mer算法可以计算基因组重复序列的大小和基因组杂合度。目前基于k-mer的研究使用的训练集数据比较大,而较长的k-mer更能表现出基因组特征的特异性。这给数据的采集、存储和处理带来了严峻的挑战,从而限制了该技术的推广和应用。可以预见的是,这些限制在未来将很快被克服,后续发展将迅速到来,提高基因组鉴定和分析的速度。本研究建立了一个新的金黄色葡萄球菌耐药预测模型,该模型基于抗菌素耐药表型和k-mer计算,结合机器学习算法,使用相对较少的训练分离株,可以准确预测抗菌药物的MICs。
本文首先分析了来自中国14个省的466株金黄色葡萄球菌分离株,包括249株耐甲氧西林金黄色葡萄球菌(MRSA)和217株甲氧西林敏感金黄色葡萄球菌(MSSA),鉴定出23例ST,其中ST59、ST239和ST398数量较高(图1),2株分离株的ST无法鉴定。抗生素敏感性测试结果表明,万古霉素、利奈唑胺和达托霉素分离株均是易感的。因此,我们分析了其他模型抗菌药物的分类性能以及可能影响分类结果的因素。本研究的工作流程如图2所示,结合随机森林、SVM和XGBoost三种机器学习算法,从全基因组测序数据中直接提取k-mer,预测了10种抗菌药物对金黄色葡萄球菌的最低抑制浓度。大多数抗菌素的基本一致性和类别一致性在两倍稀释范围内可能达到>85%和>90%(图3)。对于克林霉素、头孢西丁和甲氧苄啶-磺胺甲噁唑,基本一致性和类别一致性分别达到>91%和>93%,为临床治疗提供了重要信息。成功预测头孢西丁耐药性,表明该模型能够识别MRSA。此外,当使用全基因组测序结合k-mer和机器学习算法时,抗菌素耐药性预测比常规临床试验的检测时间快6小时。
这项研究的结果可以帮助提高临床经验性治疗的准确性,特别是在没有办法快速获得抗菌药敏结果的情况下。此外,单独使用k-mer提取基因数据特征可以与宏基因组学联系起来。在物种测定后直接对MIC进行分析可以加快未来的病原体诊断和抗菌药敏试验。
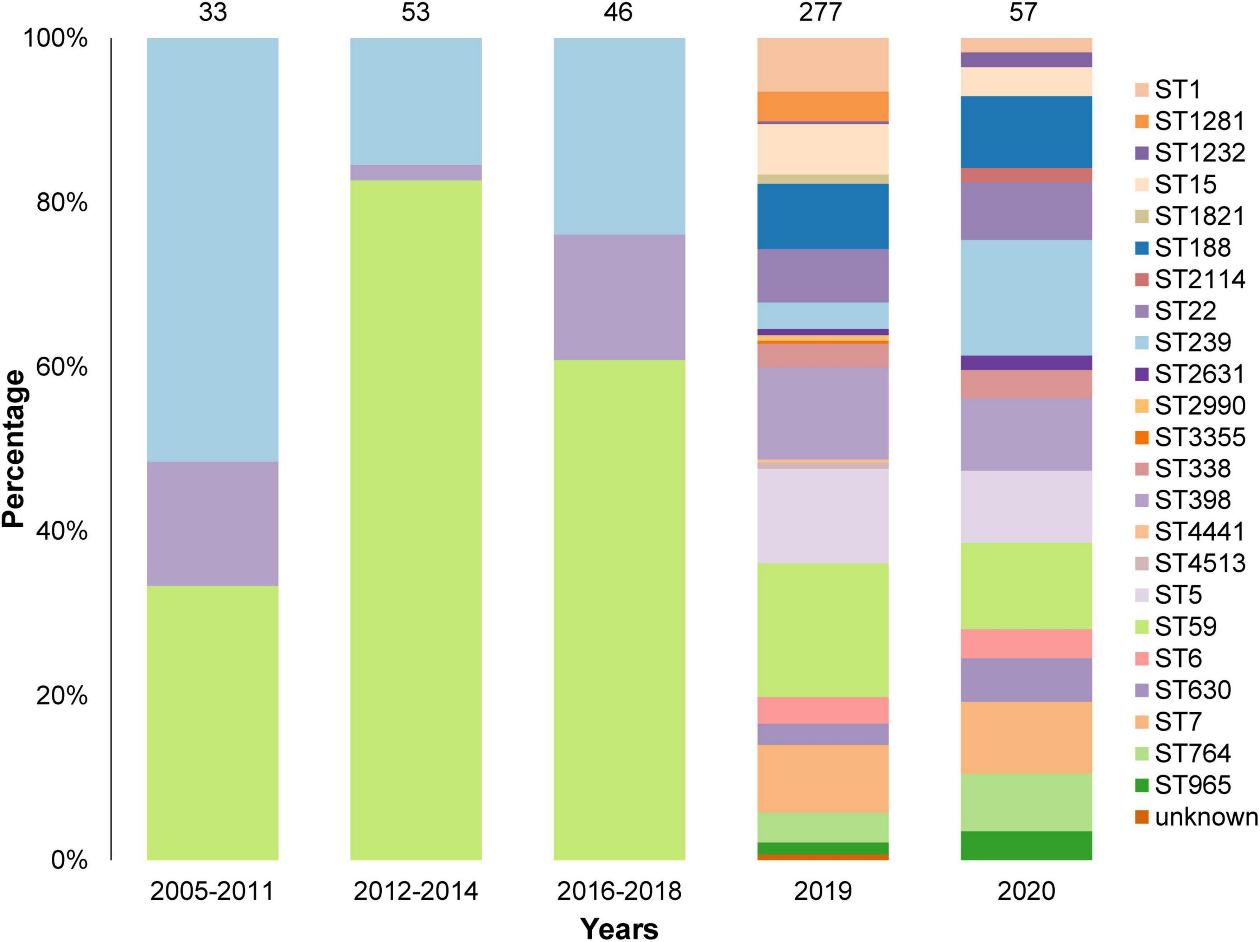
图1本研究中466株分离株的序列类型(ST)和收集年份。共鉴定出23例ST,2株分离株的ST未知。大多数分离株是在2019年和2020年收集的。
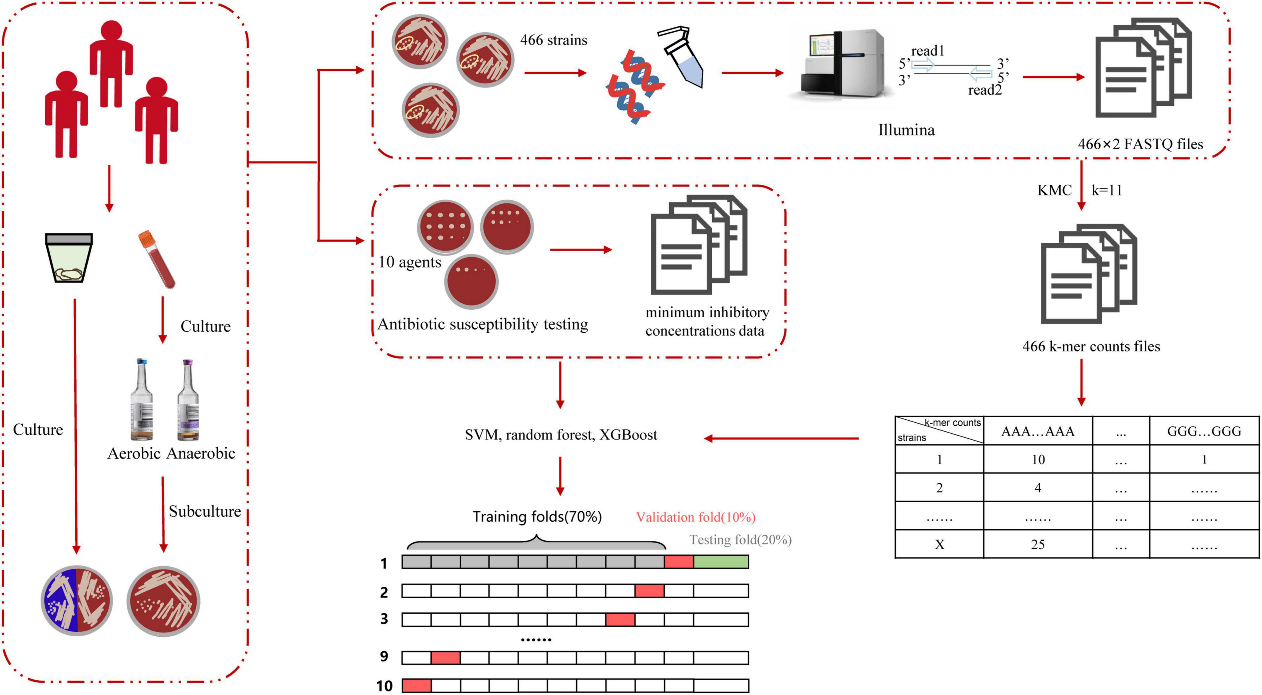
图2本研究中实际操作的工作流程示意图。从样本中提取DNA并由Illumina测序以获得FASTQ文件。使用KMC获取k-mer计数文件,并转换k-mer计数文件的格式以获得矩阵,其中行包含分离株的数量,列包含k-mer计数。最后,我们使用各种机器学习算法在测试集中学习并获得预测精度。
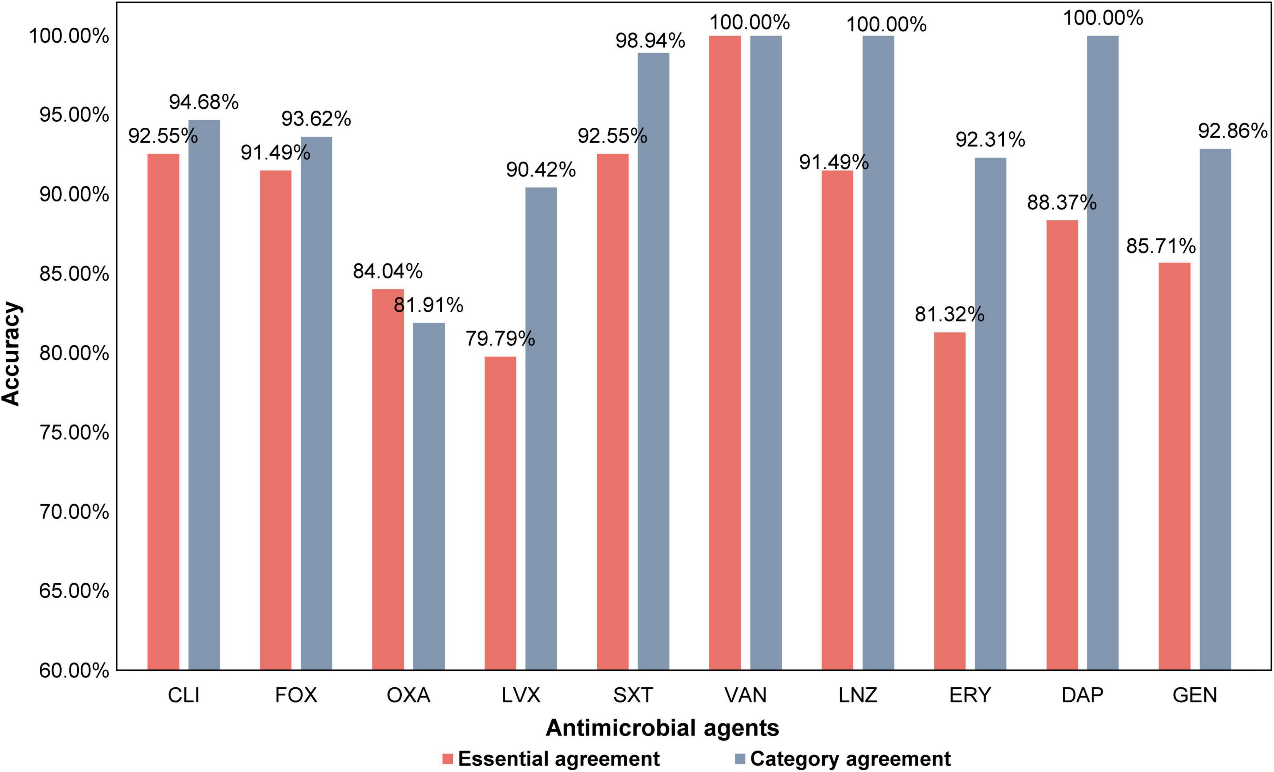
图3.所有抗菌素的基本一致性(EA)和类别一致性(CA)在两倍稀释范围内的预测精度。
参考文献:
[1]S. Wang, C. Zhao, Y. Yin, F. Chen, H. Chen, H. Wang, A Practical Approach for Predicting Antimicrobial Phenotype Resistance in Staphylococcus aureus Through Machine Learning Analysis of Genome Data, Front Microbiol, 13(2022) 841289.
链接:https://doi.org/10.3389/fmicb.2022.841289
1、凡本网所有原始/编译文章及图片、图表的版权均属微生物安全与健康网所有,未经授权,禁止转载,如需转载,请联系取得授权后转载。
2、凡本网未注明"信息来源:(微生物安全与健康网)"的信息,均来源于网络,转载的目的在于传递更多的信息,仅供网友学习参考使用并不代表本网同意观点和对真实性负责,著作权及版权归原作者所有,转载无意侵犯版权,如有侵权,请速来函告知,我们将尽快处理。
3、转载请注明:文章转载自www.mbiosh.com
联系方式:020-87680942

