Adv. Sci. |DNA片段化:淋病奈瑟菌的药敏分析新方案
Adv. Sci. |DNA片段化:淋病奈瑟菌的药敏分析新方案


AMR是全球医疗健康系统面临的重大挑战,尤其是淋病奈瑟菌(N. gonorrhoeae),它已经对所有现有的抗菌药物产生了耐药性。其中,头孢曲松(CEF)是目前治疗淋病奈瑟菌感染的最后一种经验性治疗方法。耐药菌株的出现使得淋病的治疗变得复杂,增加了治疗失败的风险。因此,快速准确地给出药物表型结果是延缓细菌耐药性发展的关键。然而,传统的基于培养的药敏测试(AST)方案通常耗时数天,而新兴的基于基因型的AST方案又因细菌耐药机制的复杂性,无法给出准确的表型结果。此外,尽管表型-基因型AST方案近年来得到了广泛关注,但淋病奈瑟菌的opa基因是多拷贝的,无法直接反映细菌的生存状态,这限制了其在药敏测试中的应用。
幸运的是,Tza-Huei Wang团队观察到淋病奈瑟菌在暴露于β-内酰胺类抗菌药物时,会出现DNA片段化的现象。基于此,该团队开发了一种新型的表型-分子型AST方案——基于opa基因和终止子(terminus)位点的双重数字PCR技术。其原理如下:opa基因在细胞内以多拷贝形式存在,而终止子位点是细菌染色体末端的一个单拷贝基因片段。在数字化系统中,完整的细胞会同时含有opa基因和终止子位点,因此在PCR扩增后,微孔中会同时检测到两种荧光信号。而细胞破裂后,微孔中通常只含有单拷贝基因(opa基因或终止子位点),因此只会产生一种荧光信号(图1)。
在实验中,作者设计了两组实验:对照组(无抗生素)和处理组(有抗生素)。通过计算两组实验的DNA与细胞的比值(Ss),并设定阈值(耐药菌的所有Ss值的平均值加上3倍标准偏差),来判断菌株的药敏性。如果Ss值大于阈值,则说明该菌株为敏感或中介;反之则为耐药。作为验证,作者分别利用10株菌株对青霉素(PEN)和8株菌株对CEF的药敏性进行了测试(图2)。
总的来说,文章通过开发双重数字PCR技术,实现了对淋病奈瑟菌的快速药敏测试。该方法利用细胞裂解和DNA片段化作为药敏评估的指标,具有高灵敏度和特异性,能够在短时间内提供可靠的药敏结果。尽管存在样本量有限、实验条件的局限性和技术复杂性等局限性,但该方法在快速诊断和临床应用中具有重要的潜力。
图1 (A)双重数字PCR的设计原理。敏感株(B)和耐药株(C)在青霉素处理1小时后的数字PCR检测结果。
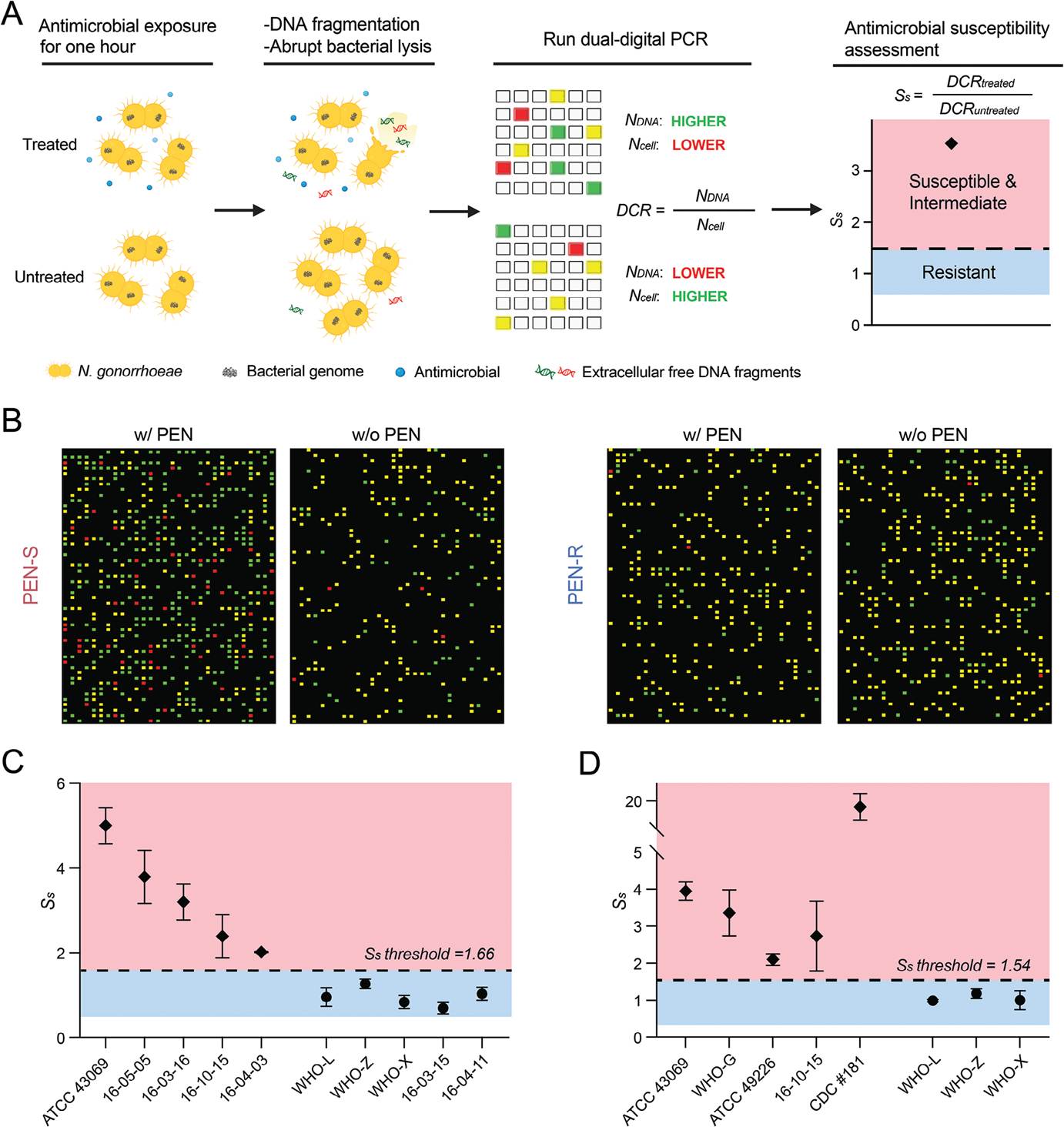
图2 (A)双重数字PCR用于淋病奈瑟菌药敏测试的工作流程。(B)PEN敏感(ATCC 43069)和耐药菌株(16-04-11)的药敏测试结果。多种菌株对PEN(C)及CEF(D)的药敏测试结果。
上一篇:新型 MALDI-TOF-MS 检测法助力艰难梭菌感染诊断
下一篇:揭秘昆虫“肠道秘密”:Cell Painting技术如何改写杀虫剂研究未来?
1、凡本网所有原始/编译文章及图片、图表的版权均属微生物安全与健康网所有,未经授权,禁止转载,如需转载,请联系取得授权后转载。
2、凡本网未注明"信息来源:(微生物安全与健康网)"的信息,均来源于网络,转载的目的在于传递更多的信息,仅供网友学习参考使用并不代表本网同意观点和对真实性负责,著作权及版权归原作者所有,转载无意侵犯版权,如有侵权,请速来函告知,我们将尽快处理。
3、转载请注明:文章转载自www.mbiosh.com
联系方式:020-87680942




