快速锁定VRE耐药性:机器学习与MALDI-MS的强强联合
快速锁定VRE耐药性:机器学习与MALDI-MS的强强联合


VRE感染的全球流行对公共卫生构成了重大威胁。VRE是一种对多种抗生素具有耐药性的细菌,其感染会导致疾病持续时间延长、死亡率上升以及医疗成本增加。传统抗生素敏感性测试(AST)通常需要长达72小时才能得出结果,这在早期诊断阶段严重延迟了抗感染治疗的实施,难以满足快速精准治疗的需求,不利于VRE感染的防控。近年来,MALDI-MS技术在微生物分析领域崭露头角。它能依据微生物独特的质谱指纹图谱,快速准确地识别微生物,且样本制备简单、检测通量高,在临床微生物学和环境监测等方面应用广泛。在AST方面,MALDI-MS也展现出巨大潜力,为快速检测VRE带来了新希望。与此同时,机器学习在生命科学领域的应用日益深入,可挖掘复杂数据中的模式、相关性及预测关系。将机器学习与MALDI-MS相结合,能够实现对数据的深度解读。基于此,Gao等人开发了一种结合MALDI-MS和机器学习的集成策略,以快速预测VRE,应对VRE感染带来的严峻挑战。
文章的主要研究内容包括三个部分(图1):
(1)图谱数据库的构建:针对从航天中心医院收集到的398株粪肠球菌样本(198株VRE和200株VSE),在培养24小时和48小时的时间点进行MALDI-MS分析,生成了包含2388个质谱图谱的数据集。
(2)机器学习建模:对质谱图谱数据进行校准、归一化等预处理后,输入四种机器学习模型:支持向量机(SVM)、SVM with L1-norm(L1-SVM)、逻辑回归(LR)和多层感知器(MLP)。通过外部验证(将数据集划分为内部数据集和外部数据集)和时间验证(将三次数据采集中的前两次作为训练集,第三次作为验证集)两种验证方式,评估模型在独立样本上的泛化能力以及在不同时间点数据上的鲁棒性。基于准确率、召回率、F1分数和AUROC值等评估参数,作者确定L1-SVM为最优模型。该模型在24小时和48小时培养的外部验证中AUROC值分别为0.84和0.88,在时间验证中分别为0.71和0.80(图2)。
(3)质谱特征识别:利用串联质谱(LC-MS/MS)对关键质谱区域中的特定峰值进行进一步分析,以识别潜在的生物标志物。通过机器学习模型识别出33个关键质谱区域,并利用串联质谱进行蛋白鉴定。结果显示,鉴定出的某些核糖体蛋白,如A0A133N352和R2Q455,可能作为区分VRE和VSE的生物标志物(图3)。
总的来说,文章联用MALDI-MS和机器学习技术,开发了一种快速预测VRE的新方法。该方法能够在24小时内提供准确的预测结果,显著提高了抗生素耐药性检测的速度和效率。通过识别关键质谱特征和潜在的生物标志物,该方法为临床快速诊断和抗生素治疗提供了重要的理论依据和实践指导。
图1 研究工作流程示意图
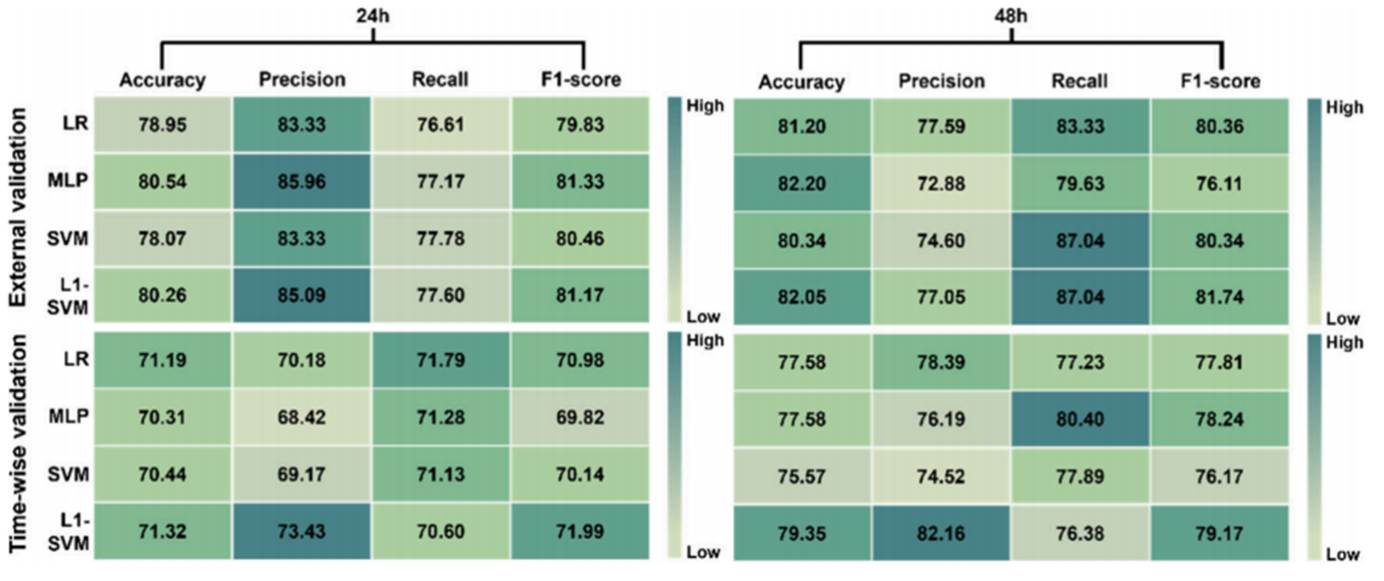
图2 四种机器学习模型针对2种数据集的预测性能
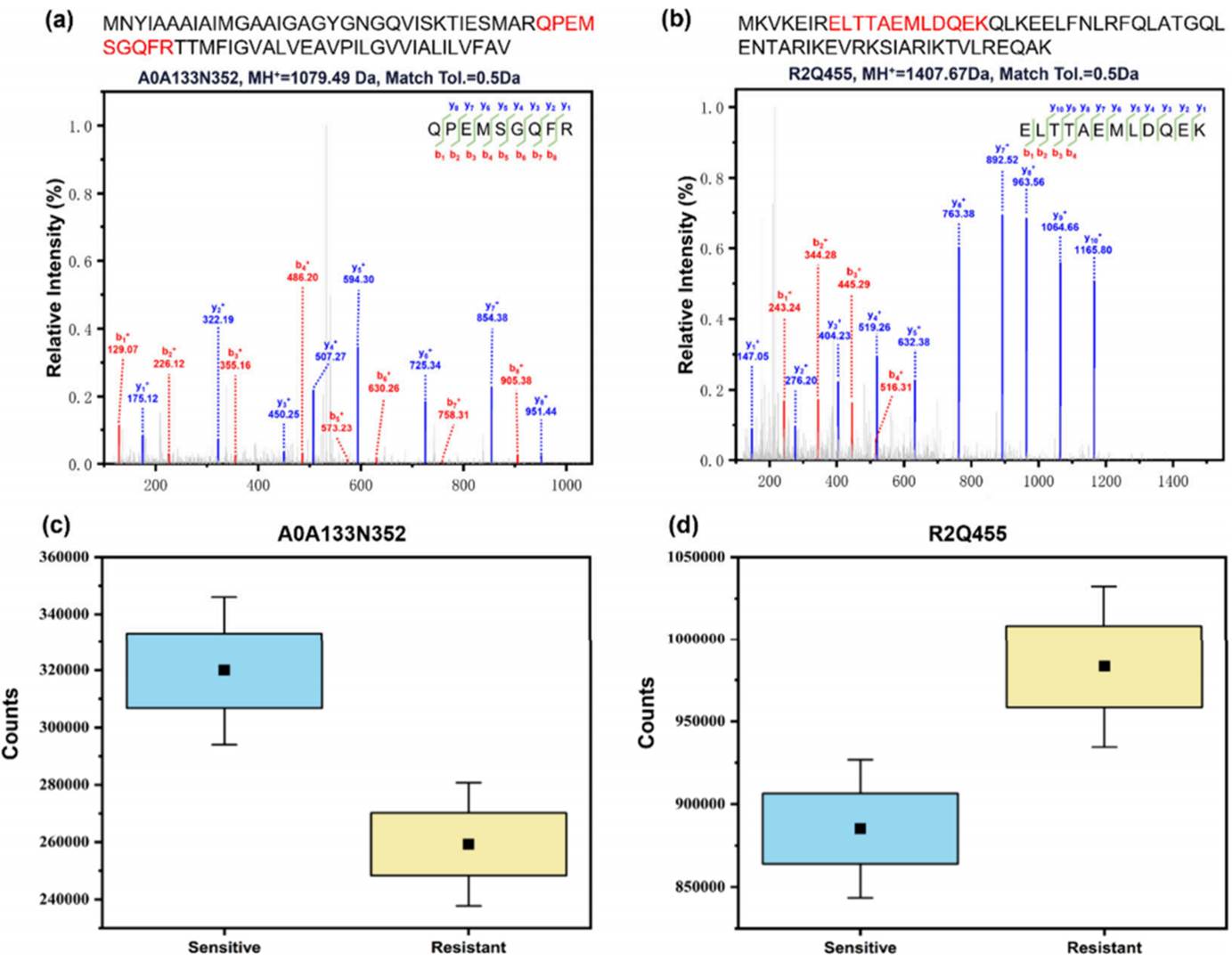
图3 通过串联质谱分析鉴定出的两个关键蛋白质(A0A133N352和R2Q455)的质谱图和表达量分析
上一篇:新型比色生物传感器:高效检测沙门氏菌,守护食品安全
下一篇:葡萄状荧光点:多病原菌检测的新突破
1、凡本网所有原始/编译文章及图片、图表的版权均属微生物安全与健康网所有,未经授权,禁止转载,如需转载,请联系取得授权后转载。
2、凡本网未注明"信息来源:(微生物安全与健康网)"的信息,均来源于网络,转载的目的在于传递更多的信息,仅供网友学习参考使用并不代表本网同意观点和对真实性负责,著作权及版权归原作者所有,转载无意侵犯版权,如有侵权,请速来函告知,我们将尽快处理。
3、转载请注明:文章转载自www.mbiosh.com
联系方式:020-87680942




