Cell Discovery|酵母中蘑菇衍生生物活性II型灵芝酸生物合成网络的解码和重编程
Cell Discovery|酵母中蘑菇衍生生物活性II型灵芝酸生物合成网络的解码和重编程


摘要
蘑菇来源的次级代谢产物具有重要的药理活性,但其生物合成途径的阐明极具挑战性,更不用说将其生物合成网络重新编程为目标代谢物。本文以传统药用真菌灵芝(Ganoderma lucidum)为例,通过研究灵芝中基因的协同表达,揭示了灵芝主要活性代谢产物II型灵芝酸(TIIGAs)的生物合成网络,鉴定了能够负责C22羟基化、C3羟基构型转化和C3、C15和C22羟基上的乙酰化的内源或异源酶。值得注意的是,我们揭示了C22羟化酶CYP512W6的催化机制,以及将乙酰基转移到C15和C22所需的意想不到的双功能乙酰转移酶GlAT。使用荧光引导的整合方法,我们实现了适用于工业发酵的重要TIIGA的高效生物合成。在将所有鉴定的酶引入工程酵母后,我们观察到下游TIIGA的生物合成受到严重阻碍,并且通过暂时调节乙酰转移酶的表达来疏通代谢阻断。通过重新编程TIIGA的生物合成网络,我们能够生产超过30种TIIGA,其滴度或效率比灵芝栽培高出1-4个数量级。这项工作使人们能够获得有价值的TIIGA,促进其广泛应用,并为其他蘑菇产品的研究提供了启示。
杂志名:Cell Discovery
英文题目:Decoding and reprogramming of the biosynthetic networks of mushroom-derived bioactive type II ganoderic acids in yeast
作者:Qin Wang,YeLi, Shun han Zhang, Wei Yuan , Ze qian Du, Ting Shi, Zhao Chang, Xing ye Zhai, Yin hua Lu, Meng Wang, Juan Guo, Jian-Jiang Zhong and Han Xiao
通讯作者单位:上海交通大学
内容
1. 缩小后修饰基因的候选范围
使用灵芝菌丝体粗蛋白裂解物,以GA-Jb作为底物进行体外活性测定。液相色谱-质谱(LC-MS)分析显示,生成了化合物1,表明,C3羟基的构型转化可以通过两步反应来完成,即C3羟基的氧化,然后C3酮基的还原。采用蓝光暴露策略来特异性刺激菌丝体中TIIGA的产生,随后进行RNA测序。使用CYP5150L8和CYP512W2作为共表达分析的诱饵基因,发现168个转录物与它们的表达高度相关。基于与TIIGA的底物结构相似性,最终选择了12种CYP、8种AT和11种脱氢酶/还原酶。
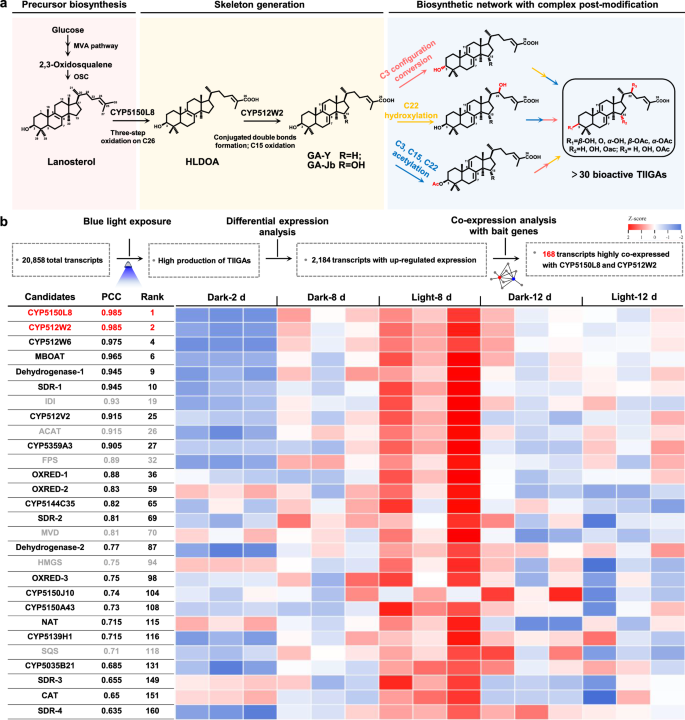
图1 灵芝中TIIGAs的生物合成网络分析
2. C3羟基构型转化的异源氧化酶和还原酶的鉴定
为了鉴定负责C3羟基构型转化的酶,将异源脱氢酶候选物引入到GA-Jb生产菌,烟曲霉(AfuSDR)和甜橙(CsSDR)的短链脱氢酶/还原酶能够催化GA-Jb的转化以产生GA-TR(图2a)。来自智人的AKR1C2和AKR1C4以及来自纺锤枝顶孢霉的FusC1能够将GA-TR完全转化为GA-Ja(图2b)。将两种氧化酶(CsSDR和AfuSDR)和三种还原酶(AKR1C2、AKR1C4和FusC1)随机分组为六种氧化酶-还原酶对,并将它们与CYP512W2一起引入HLDOA生产酵母中。CsSDR和AKR1C4的共表达产生33.3mg/L GA-Ja并积累5.4mg/L GA-Jb,没有可检测的GA-TR(图2c)。C3羟基的氧化还原介导的构型转化可以应用于其他GA,但其对HLDOA和GA-Y的C3羟基的转化不如GA-Jb有效。
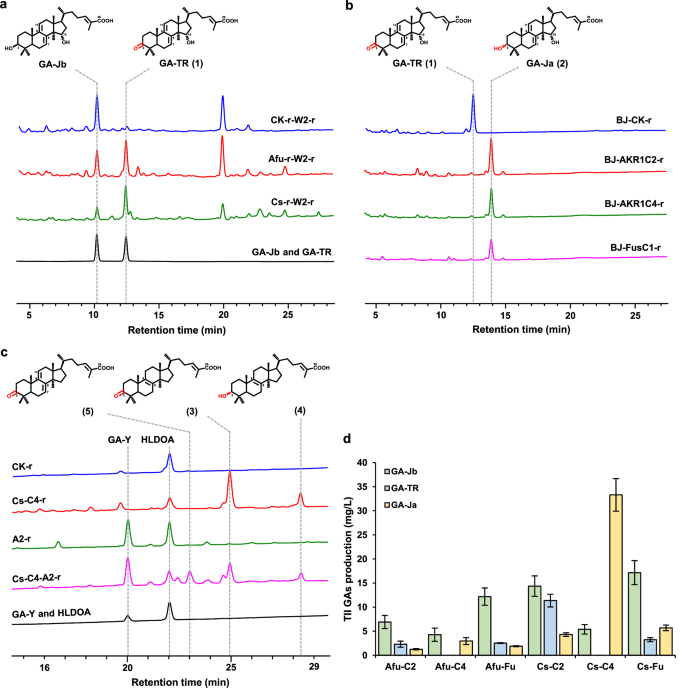
图2 C3氧化酶和还原酶的功能特性
3. CYP512W6作为C22羟化酶的鉴定及其催化机制的阐明
体内异源表达发现CYP512W6可以将HLDOA、GA-Y和GA-Jb的C22位羟基化。酵母微粒体体外反应验证CYP512W6只能催化GA-Jb上的C22羟基化,而不能催化其他GA上的C22羟基化,GA-Jb是CYP512W6的最佳底物(图3a,b,c,d)。为了研究CYP512W6介导的GA-Jb上C22羟基化的催化机制,通过AlphaFold 2对CYP512W6的结构进行建模。发现除了催化口袋周围残基的变化外,CYP512W6和CYP512W2的总体结构相似。量子力学(QM)计算和分子动力学(MD)模拟共同证明GA-Jb可以被CYP512W6羟基化(图3e)。基于MD模拟,我们鉴定了8个残基(V105、L108、L109、T212、S363、L364、M365、R366),结构分析表明,T212的侧链和M365的主链分别与GA-Jb的C3羟基和C26羧基形成氢键,而V105、L108和M365则与GA-Jb的四环发生疏水作用(图3g)。此外,S363与GA-Jb的C15羟基形成硫-氢键,R366与血红素的羧基形成盐桥。设计五个突变体(V105 L,L108 A,T212 A,M365 A和R366 A),发现所有突变体都表现出对GA-Jb的活性显著降低(图3h)。
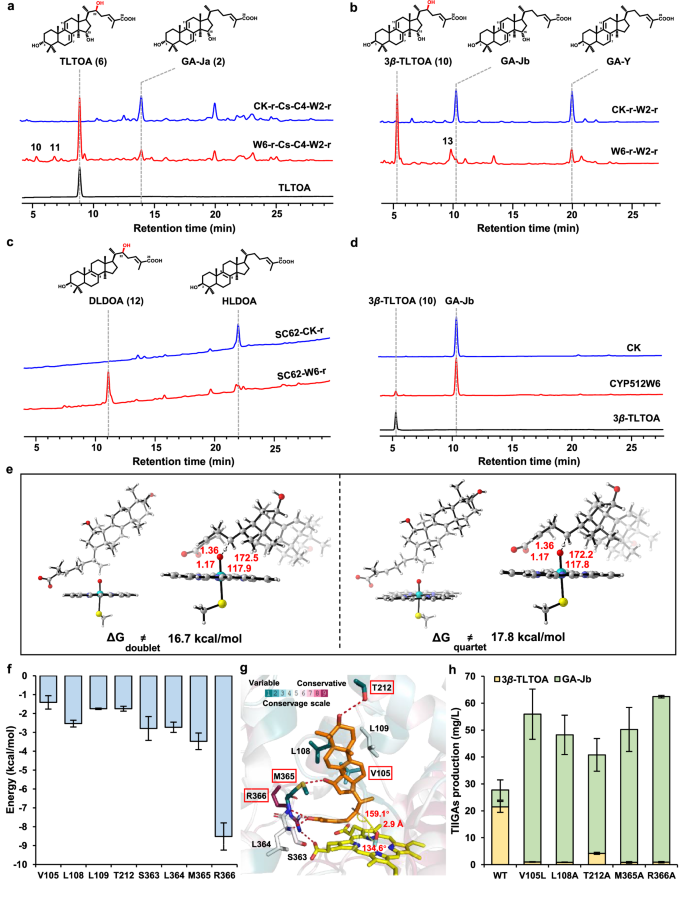
图3 CYP512W6作为C22羟化酶的发现
4.GlAT作为C15和C22羟基上双功能乙酰转移酶的鉴定及其催化机制的阐明
体内异源表达GlAT,以GA-Jb和GA-Ja为底物,检测到C15乙酰化的产物。制备GlAT的酵母微粒体,并将它们与不同的底物一起孵育。发现它可以催化各种底物的C15羟基的乙酰化。表明GlAT是能够在C15和C22上乙酰化的双功能乙酰转移酶。使用AF2对GlAT进行结构预测。对接结果表明,GA-Jb的O 1与酰基辅酶A的Cα之间的距离以及H305的Nε与GA-Jb的O 1之间的距离均为3.1 nm,适合乙酰化(图5 c)。MD模拟结果证明了GlAT在C15和C22位置转移酰基的能力。根据MD模拟结果,观察到T型隧道周围的G208和A211为GA-T2入口提供空间。S79和R218与GA-T2尾部的相互作用影响乙酰化位置(图5 b)。因此,S79 L、G208 L、A211 L、A211 F和R218 L被设计为改变T-通道大小或破坏相互作用以改变酰化位点。
H305 A在维持C15和C22上的乙酰化活性方面的关键作用。A211可能是双功能乙酰化过程的另一个重要残基,G208可能负责维持C22乙酰化活性。一种可能的解释是,208位的亮氨酸比同一位置的甘氨酸产生更大的空间位阻。这种空间位阻的差异不同地影响具有不同结构特征的底物,导致不同底物在相同化学位置处的不同选择性。推断双官能乙酰化活性可能受到C3、C15和/或C22上的全面修饰的影响。A211 F突变可能完全阻断了Ttunnel,阻止底物进入催化位点,而A211 L突变虽然也减少了T-tunnel空间,但在接近催化位点时仍为GA-T2的C22乙酰化留下了足够的空间。然而,剩余的空间不足以使C15乙酰化所需的进一步向内移动。
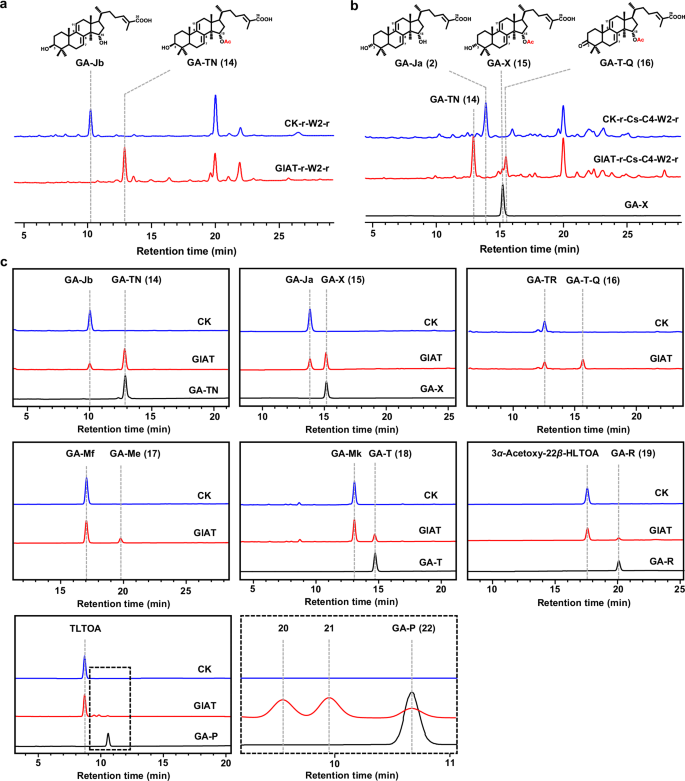
图4 GlAT的功能表征
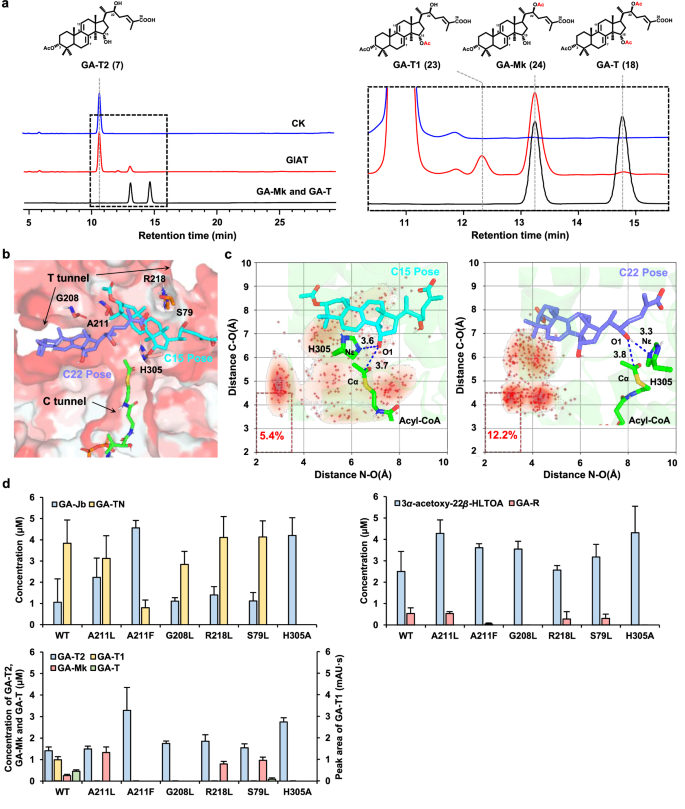
图5发现GlAT作为C15和C22上的双功能乙酰转移酶
5.异源BsAT作为C3羟基乙酰转移酶的鉴定
异源表达证明BsAT能够乙酰化TIIGA的不同C3羟基构型。BsAT对其他TIIGA(DLTOA、GA-S、TLTOA、GA-P)具有催化活性,而不管后修饰(羟基化或乙酰化)是否发生在C15和/或C22位置。BsAT具有底物偏好性,GA-S是最有利的,能够完全转化。与C3β-羟基底物相比,BsAT对C3α-羟基底物具有较高的催化活性,对C15-乙酰化TIIGA和C22-羟基化TIIGA具有较高的转化效率。
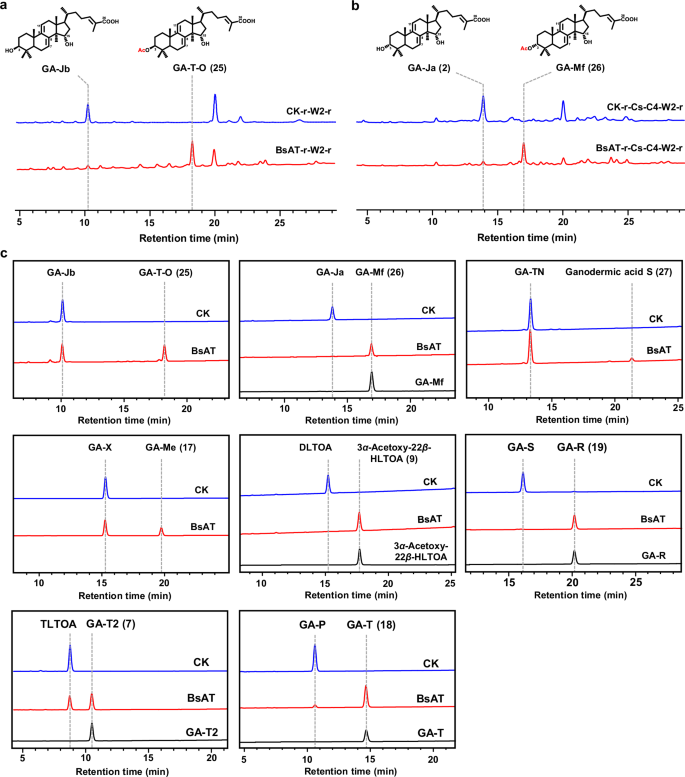
图6 BsAT的功能表征
6. 重编程TIIGAs的生物合成网络
通过荧光引导整合技术将催化模块整合进酵母基因组。并通过荧光激活细胞分选(FACS)进行筛选(图7a)。在不添加抗生素的情况下,菌株SC27能够产生最高量的GA-Jb,为39.6 mg/L(图7 b)。将其进一步在含有1g/L 5-FOA的YPD培养基中进行培养以回收CRISPR质粒。最终得到的菌株SC27*能够在120小时发酵后产生41.1 mg/L的GA-Jb(图8)。
将GA-Jb生产菌株SC27*工程化以生产GA-Ja和TLTOA。菌株SC 3和SC 1能够在120小时发酵后分别产生最高量的GA-Ja(87.5mg/L)和TLTOA(37.4mg/L),而不添加抗生素。经质粒回收后,最终得到的菌株SC 3 * 和SC 1 * 在发酵120 h后分别能够产生47.0 mg/L的GA-Ja和29.2 mg/L的TLTOA。
通过使用诱导型启动子来驱动BsAT和/或GlAT的表达来控制后修饰反应的顺序。选择半乳糖诱导型启动子PGAL1来驱动BsAT基因表达,促进C3位的乙酰化反应。采用了PADH2(乙醇诱导型启动子)和PGAL1来控制GlAT的表达,延缓C15与C22位乙酰化反应,以积累中间产物GA-T。最终通过工程酵母菌株的改造,超过30种TIIGA的生产效率或产量比来自养殖蘑菇的生产效率或产量高1-4个数量级。
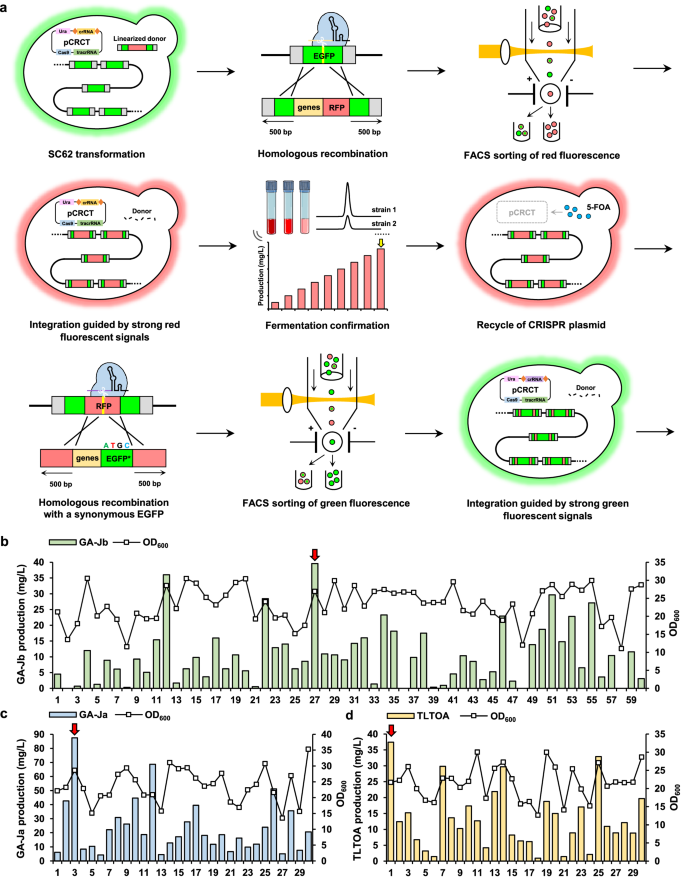
图7关键酶表达盒的整合导致TIIGA的有效产生
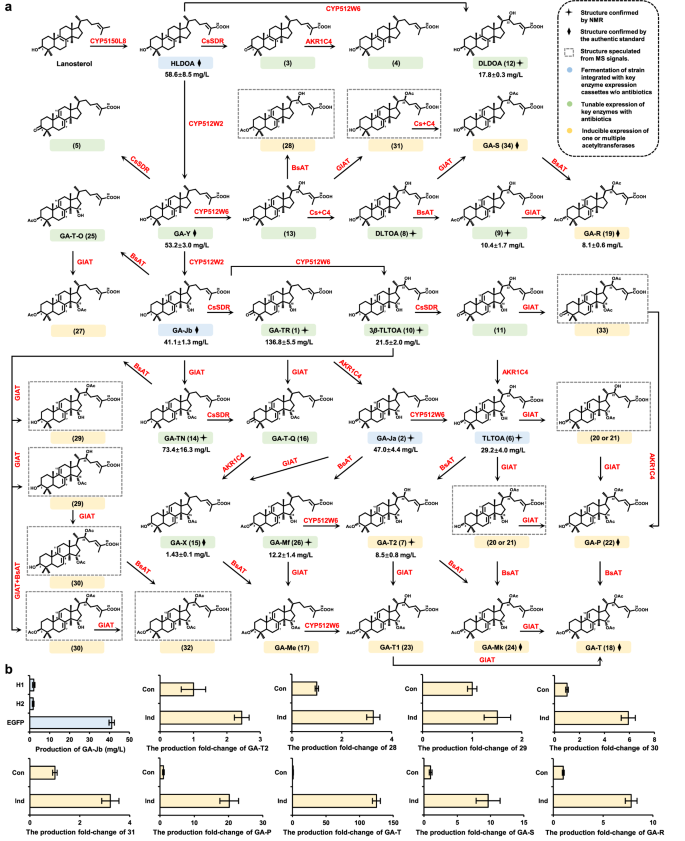
图8将生物合成网络重编程为靶向TIIGA
上一篇:羊肚菌科真菌生态生活方式与基因组演化研究获新进展
下一篇:科研进展 | 四川省食用菌所在光质调控粉色木耳生长及氨基酸代谢研究取得新进展



